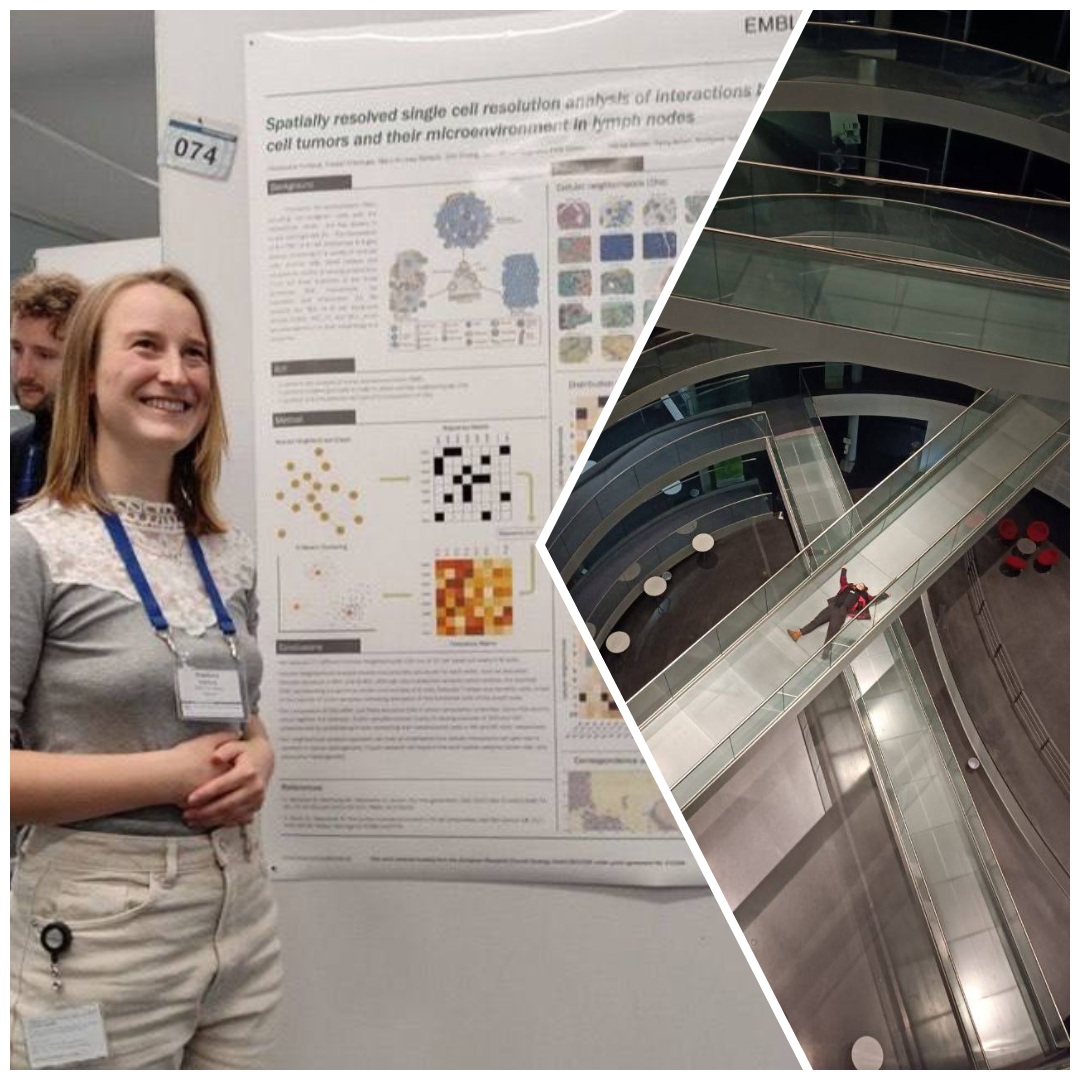
Публікуємо #HumansOfGenomicsUA з учасницею спільноти Анастасією Горловою (LinkedIn).
Привіт усім, мене звати Настя. Зараз я живу в Німеччині, але мої друзі жартують, що справжньої Німеччини я не бачила. Чому так? Бо працюю я у великому інтернаціональному інституті, де вітаються люди всіх національностей та підтримується мультикультурність. Інститут цей — EMBL (European Molecular Biology Laboratory) Heidelberg.
У Гайдельберзі знаходиться головна філія інституту, і ще пʼять розташовані у різних містах Європи та Англії. Разом вони забезпечують блискучі умови (матеріальні та нематеріальні) для існування якісної науки. Усі філії надають інструменти та сервіси для:
- структурної біології: electron microscopy and light microscopy facilities;
- мультиоміки: genomics facilities, proteomics facilities, metabolomics facilities, flow cytometry facilities;
- важких компʼютерних обчислень: high performance cluster;
- тощо-тощо-тощо…
Усе обладнання доступне для ваших експериментів. Для співробітників, охочих працювати за інструментом, проводять якісне навчання.
У EMBL знайти собі місце може будь-хто: біологи, хіміки, фізики, інженери та спеціалісти інших галузей. Усе це велике комʼюніті працює над тим, аби відкривати нові горизонти у сферах геноміки, протеоміки, біології розвитку, клітинної та системної біології (Гайдельберг), структурної біології (Гайдельберг, Гамбург, Гренобль), нейробіології та епігенетики (Рим), біології тканини та моделювання захворювань (Барселона), біоінформатики (Гайдельберг, Хінкстон) та в багатьох інших дотичних галузях.
А ще, окрім неймовірної науки, EMBL вирізняється атмосферою та способом взаємодії. Ти будуєш дружні стосунки не лише зі своїми однолітками, а й зі старшими колегами, також можеш вільно спілкуватися з очільником будь-якої лаби й не відчувати барʼєру та напруження стосовно “статусу” співрозмовника.
Оскільки я базуюсь в Гайдельберзі, то можу розказати трохи більше саме про це місце. EMBL тут — це величезний модерний кампус на схилі, оточений зеленим лісом, тут найкраща їжа в їдальні (я не збрешу, якщо скажу, що на рівні хорошого ресторану), приємні лаунж-зони для співробітників, періодичні вечірки, а також безкоштовне пиво з піцою щопʼятниці для заохочення нетворкінгу 😉
Одна з тих речей, що подобаються мені найбільше стосовно EMBL у Гайдельберзі — це ATC (Advanced Training Center), де щорічно проводиться купа конференцій. Якщо ти знаходишся в кампусі на час проведення чергової крутої події, можна прийти і послухати провідних науковців світу, а після їх виступу побалакати з ними за кавою.
Потрапити в це середовище можна на стажування, PhD, Postdoc або на будь-яку іншу відкриту позицію. Стажування можливе завдяки написанню листів очільникам лабораторій, які вас цікавлять. Багатьом з них потрібні люди для численних проєктів. Багато з них мають фінансування, тож за стажування можуть платити гроші. Часто це зарплата, яка відповідає прожитковому мінімуму. Онлайн стажування також можливі, особливо, якщо ви займаєтеся біоінформатикою. Щодо PhD, то в EMBL існує ціла програма для щорічного рекрутингу студентів — EIPP. Дуже рекомендую її спробувати, якщо PhD — це те, що вас цікавить. Отож не соромтеся написати email у лабу вашої мрії — імовірність туди потрапити існує завжди 😉
Я була б рада вітати в EMBL більше гостей з України, і було би класно, якби наше представництво тут росло та зміцнювалося. Про всі-всі плюшки EMBL у малому повідомленні не розповіси, тож залюбки відповім на ваші додаткові запитання 😉
Нам цікаво дізнатися чим живе наша спільнота. Є чим поділитися про себе, що могло б допомогти або надихнути інших на їх кар’єрному шляху? Напишіть сюди: team-genomics@genomics.org.ua і давайте поспілкуємося про публікацію.